6.2 Organismos eucarióticos
La estructura de los ARNm y el proceso de transcripción en las células eucariotas, es similar a lo expresado anteriormente para las células procariotas. Sin embargo, debemos considerar las siguientes diferencias.
a) Las células eucariotas poseen tres clases de ARN polimerasas (I, II y III), las cuales se utilizan para sintetizar los distintos tipos de ARN existentes.
b) Los extremos 3’ y 5´ de los ARNm están modificados. En el caso del extremo 5’ encontraremos una estructura denominada CAP y, en el correspondiente extremo 3’, se encuentra adherido una larga secuencia de nucleótidos cuya base nitrogenada es la Adenina (Cola Poly A)
c) Las moléculas de ARNm, luego de ser sintetizas, son modificadas. Los “transcriptos primarios” eucariotas sufren un proceso por el cual determinadas secuencias (Intrones) son eliminadas.
d) Los ARNm eucariotas son Monocistrónicos.
En las células del tipo Eucariota, encontramos tres tipos de ARN polimerasa las cuales se denominan: ARN polimerasa I, ARN polimerasa II y ARN polimerasa III. Sus composiciones en sub unidades son complejas y aun no se conoce en exactitud la función de cada una de estas sub unidades. Lo que sí se conoce con exactitud, es la localización y el producto de cada una de ellas.
La mayoría de los promotores tienen una secuencia denominada TATA box o Caja Hogness, centrada a –25 Pb del punto de inicio. Esta TATA box es idéntica a la mencionada en la Transcripción de las células Procariotas, variando solamente en su localización.
Los promotores de la ARN Polimerasa II muestran una mayor variación en secuencia y la enzima es incapaz de iniciar la transcripción sola, sino que requiere de una serie de factores de transcripción, los cuales son los encargados del reconocimiento de un promotor particular. Recordemos que, en las células Procariotas, el factor sigma es el que le confiere a la ARN Polimerasa la capacidad de seleccionar a cada uno de los promotores.
Los promotores Eucariotas no trabajan solos para asegurar una transcripción eficaz. En algunos genes o tipos celulares, la actividad de un promotor está aumentada por la presencia de otra secuencia conocida como Exaltador o Enhacer.
Si bien la síntesis y estructura de los ARN mensajeros en Eucariotas es similar a las
Procariotas, deben mencionarse las siguientes diferencias:
a) Ambos extremos están modificados, el 5’ presenta una estructura denominada CAP (caperuza) y el 3’ contiene una secuencia de Ácido Poliadenílico denominada Cola
Poly A.
b) La molécula que sirve de molde para la síntesis de proteína (ARNm maduro) es más corta que el ARN mensajero recién sintetizado (Pre – ARN mensajero). Esto se debe a que los últimos sufren un proceso de eliminación de secuencias no codificantes. Dicho proceso se denomina Splicing.
Diferencias entre la transcripción procarióticas y eucarióticas
1._ En los procariotas el ARNm no tiene ni caperuza ni cola.
2._ Tampoco tiene intrones y por lo tanto no requiere de un mecanismo de maduración.
3._ Al mismo tiempo que el ARNm se transcribe se está ya traduciendo.
4._ Los genes son policistrónicos, esto es, un ARNm contienen información para varias proteínas.
5._ En los eucariotas, la transcripción y la traducción no están acopladas, y se producen en compartimentos diferentes. Esto implica una regulación distinta para cada proceso.
En las células del tipo Eucariota, encontramos tres tipos de ARN polimerasa las cuales se denominan: ARN polimerasa I, ARN polimerasa II y ARN polimerasa III. Sus composiciones en sub unidades son complejas y aun no se conoce en exactitud la función de cada una de estas sub unidades. Lo que sí se conoce con exactitud, es la localización y el producto de cada una de ellas.
La RNA-polimerasa I se localiza en el nucléolo por ser donde se transcriben activamente los rRNA. Es muy abundante y es la más activa. La cromatina que transcribe está totalmente desprovista de nucleosomas para permitir la transcripción rápida y continua de estos genes.
La RNA-polimerasa II transcribe todos los genes que se traducen y los snRNA de tipo U (menos el U6 que lo hace la RNA-polimerasa III), por lo que se considera la más representativa y es a la que se suele aludir cuando no se especifica el tipo de polimerasa. Es menos activa que la RNA-polimerasa I porque debe eliminar los nucleosomas a su paso.
La RNA-polimerasa III es la más compleja y menos abundante, y transcribe todo tipo de RNA pequeños.
6.2.1 Etapas de síntesis del ARN.
1. Iniciación: Una ARN-polimerasa comienza la síntesis del precursor del ARN a partir de unas señales de iniciación "secuencias de consenso " que se encuentran en el ADN
2. Alargamiento: La síntesis de la cadena continúa en dirección 5'-3'. Después de 30 nucleótidos se le añade al ARN una cabeza (caperuza o líder) de metil-GTP en el extremo 5'. Esta cabeza parece tener una función protectora para que las enzimas exonucleasas que destruyen los ARN no lo ataquen. Una vez que esto ha ocurrido, continúa la síntesis del ARN en dirección 5-3´
3. Finalización: Una vez que la enzima (ARN polimerasa) llega a la región terminadora del gen, finaliza la síntesis del ARN. Entonces, una poli A-polimerasa añade una serie de nucleótidos con adenina, la cola poli A, y el ARN, llamado ahora ARNm precursor, se libera.
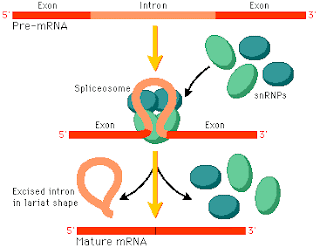
4. Maduración: El ARNm precursor contiene tanto exones como intrones. Se trata, por lo tanto, de un ARNm no apto para que la información que contiene sea traducida y se sintetice la correspondiente molécula proteica. En el proceso de maduración un sistema enzimático reconoce, corta y retira los intrones y las ARN-ligasas unen los exones, formándose el ARNm maduro.
Todo esto se ha producido en el núcleo celular. El ARNm maduro, que a partir de ahora será simplemente el ARNm o, también, el transcrito, pasará al hialoplasma donde su información servirá para la síntesis de una proteína concreta. Esto es, la información que se encuentra en forma de una cadena de nucleótidos se traducirá a una cadena de aminoácidos.
6.2.2 Modificaciones postranscripcionales del RNA mensajero
Los procesos de maduración son los que llevan a los transcritos primarios a convertirse en transcritos maduras. Mostrados sobre un mRNA, son:
Splicing: Todos los Pre- ARN m sufren un proceso denominado Splicing, que tiene como finalidad eliminar a los Intrones (secuencias no codificantes). Este proceso ocurre en el núcleo y da origen al ARN m que será desplazado al citoplasma, para ser partícipe del proceso de Traducción. En el Splicing son descartados del 50% al 90% del Pre-ARNm (transcripto primario). Los elementos remanentes (exones) son unidos o ensamblados. El número de intrones, generalmente sobrepasa al de los Exones.
Cap: El extremo 5’ contiene dos nucleótidos conectados que siempre están metilados (-CH3). La reacción de la incorporación del CAP ocurre inmediatamente después de iniciada la transcripción y siempre precede a cualquier modificación que se le realice al ARN m precursor. El CAP tendría la función de proteger al ARNm de la degradación, con lo cual aumenta su vida media.
Cola Poly A: En el extremo 3’, según se mencionó antes, los ARNm Eucariotas contienen una secuencia de 20 a 200 nucleótidos que poseen como base nitrogenada a la Adenina. La secuencia Poly A no está codificada en el ADN, sino que es añadida al ARN después de finalizada la transcripción. La función de la Cola Poly A es la de aumentar la estabilidad de los ARN m.
Regulación de la Transcripción
Dentro del metabolismo celular, encontraremos enzimas que son necesarias en forma continua y otras que solo se necesitan en determinadas circunstancias. Las primeras se sintetizan siempre, mientras que las segundas solo son sintetizadas en determinadas condiciones. Es decir que, la célula, debe contar con mecanismos que le permitan regular esta síntesis, ya que sería un gasto de energía innecesario el transcribir y traducir una proteína que luego no ha de ser utilizada.
Los principales mecanismos de regulación de la expresión génica, recaen en el proceso de transcripción. Es así como, la célula podrá “elegir” qué genes transcribe y cuáles no, dependiendo de las necesidades metabólicas reinantes en ese momento. Cada día se postulan más mecanismos por los cuales la transcripción puede ser regulada, nosotros nos dedicaremos al estudio del mecanismo clásico de regulación: el Modelo del Operón.
El Modelo del Operón fue descripto en células Procariotas, por los investigadores franceses Francois Jacob y Jacques Monod, quienes se hicieron acreedores del Premio Nobel en el año 1965.
Tal como postularan Jacob y Monod, los grupos de genes que codifican para proteínas asociadas se encuentran en unidades conocidas como Operones. Un Operón está constituido por: un Promotor, un Operador y Genes Estructurales. El Promotor, según vimos en el apartado anterior, es el lugar del ADN en el cual se une la ARN polimerasa para iniciar la transcripción. El operador es una secuencia de nucleótidos que se encuentra interpuesto entre el promotor y los genes estructurales. Los Genes Estructurales son las porciones de ADN que codifican para la síntesis de las proteínas asociadas metabólicamente.
La transcripción de los Genes Estructurales depende de la actividad de otro gen denominado Gen Regulador, que generalmente esta ubicado a unos cientos de pares de bases del sitio promotor. El gen Regulador codifica para una proteína que tiene afinidad para unirse al operador. Siempre que el represor se encuentre unido a esta proteína, se verá boqueada la unión de la ARN polimerasa al promotor, inhibiéndose así la transcripción de los Genes estructurales.






No hay comentarios:
Publicar un comentario